Anticorpi umani ultrapotenti proteggono dalla sfida di SARS-CoV-2 tramite più meccanismi
Lo studio su Science :
Opzioni terapeutiche efficaci sono necessari per controllare la diffusione della SARS-CoV-2 che ha causato più di 922.000 morti a partire dal 13 settembre °, 2020. Riportiamo l’isolamento e la caratterizzazione di due anticorpi neutralizzanti umani SARS-CoV-2 ultrapotenti (S2E12 e S2M11) che proteggono i criceti dalla infezione della SARS-CoV-2. Le strutture di microscopia crioelettronica mostrano che S2E12 e S2M11 bloccano in modo competitivo l’attacco ACE2 e che S2M11 blocca anche il picco in una conformazione chiusa mediante il riconoscimento di un epitopo quaternario che attraversa due domini adiacenti di legame del recettore. I cocktail che includono S2M11, S2E12 o l’anticorpo S309, precedentemente identificato, neutralizzano ampiamente SARS-CoV-2 circolanti e attivano le funzioni effettrici. I nostri risultati aprono la strada all’implementazione di cocktail di anticorpi per la profilassi o la terapia, aggirando o limitando l’emergere di virus mutanti.
Opzioni terapeutiche efficaci sono necessari per controllare la diffusione della SARS-CoV-2 che ha causato più di 922.000 morti a partire dal 13 settembre °, 2020. Riportiamo l’isolamento e la caratterizzazione di due anticorpi neutralizzanti umani SARS-CoV-2 ultrapotenti (S2E12 e S2M11) che proteggono i criceti dalla sfida della SARS-CoV-2. Le strutture di microscopia crioelettronica mostrano che S2E12 e S2M11 bloccano in modo competitivo l’attacco ACE2 e che S2M11 blocca anche il picco in una conformazione chiusa mediante il riconoscimento di un epitopo quaternario che attraversa due domini adiacenti di legame del recettore. I cocktail che includono S2M11, S2E12 o l’anticorpo S309 precedentemente identificato neutralizzano ampiamente un pannello di isolati di SARS-CoV-2 circolanti e attivano le funzioni effettrici. I nostri risultati aprono la strada all’implementazione di cocktail di anticorpi per la profilassi o la terapia, aggirando o limitando l’emergere di virus mutanti .
La sindrome respiratoria acuta grave Coronavirus 2 (SARS-CoV-2) è emersa alla fine del 2019 ed è stata sequenziata entro gennaio 2020 ( 1 , 2 ). Sebbene l’ospite del serbatoio responsabile della ricaduta nella popolazione umana rimanga incerto, SARS-CoV-2 sembra aver avuto origine in pipistrelli da cui sono stati identificati virus e sequenze virali strettamente correlati ( 1 , 3 ). SARS-CoV-2 appartiene al sottogenere del sarbecovirus ed è strettamente correlato al SARS-CoV, responsabile di un’epidemia nel 2002-2003 che ha provocato 8.098 casi e 774 decessi in tutto il mondo ( 4 , 5). La mancanza di immunità preesistente alla SARS-CoV-2 a causa della sua divergenza dai quattro coronavirus endemici circolanti e la sua elevata trasmissibilità da uomo a uomo ha portato alla pandemia COVID-19 in corso che ha già causato oltre 29 milioni infezioni e oltre 922.000 decessi a metà settembre 2020.
L’infezione da SARS-CoV-2 inizia con l’attaccamento della glicoproteina virale transmembrana spike (S) tramite un motivo di legame del recettore (RBM) all’enzima di conversione dell’angiotensina 2 (ACE2), che porta alla fusione della membrana e all’ingresso nelle cellule ospiti ( 6 – 13 ). Come per tutti i coronavirus, SARS-CoV-2 S è l’obiettivo principale degli anticorpi neutralizzanti (Abs) e un fulcro della progettazione del vaccino e degli sforzi di targeting terapeutico ( 14 ). Anche se i programmi di sviluppo di vaccini sono una corsia preferenziale ( 15 – 20), la produzione e l’amministrazione su larga scala per una popolazione sufficientemente ampia da ottenere la protezione della comunità richiederanno probabilmente molti mesi. I farmaci antivirali profilattici e / o terapeutici potrebbero colmare il divario prima che vaccini sicuri ed efficienti diventino ampiamente disponibili e continueranno ad avere utilità in individui non vaccinati o in coloro che rispondono male alla vaccinazione.
Abbiamo recentemente descritto un Ab monoclonale (mAb), isolato dalle cellule B della memoria di un sopravvissuto alla SARS ottenuto 10 anni dopo il recupero, che neutralizza SARS-CoV-2 e SARS-CoV attraverso il riconoscimento del dominio di legame del recettore S (RBD) ma senza bloccare l’attacco ACE2 ( 21 ). Una versione ottimizzata di S309 è attualmente in fase di valutazione in studi clinici di fase 2/3. L’isolamento di molti altri RBD mirati neutralizzando Abs da COVID-19 pazienti convalescenti ( 22 – 28 ) e la dimostrazione che essi forniscono nella protezione vivo contro la SARS-CoV-2 sfida nei piccoli animali e primati non umani ( 25 , 29 – 31) hanno dimostrato che l’RBD è l’obiettivo principale della neutralizzazione degli addominali in caso di infezione da CoV naturale. La valutazione clinica di Abs terapeutico interferire direttamente con il legame ACE2 sono in corso ( il 30 – 34 ). Gli mAb con una potenza di neutralizzazione eccezionalmente elevata, insieme a meccanismi d’azione distinti e complementari rispetto agli mAb esistenti, possono consentire la formulazione di cocktail mAb con maggiore efficacia per controllare la diffusione del virus e prevenire la resistenza. Qui, abbiamo valutato la possibilità di combinare due Abs neutralizzanti ultrapotenti che abbiamo scoperto, ovvero S2E12 e S2M11, che sfruttano diversi meccanismi di azione.
Risultati
Isolamento di Abs neutralizzante SARS-CoV-2 ultrapotente
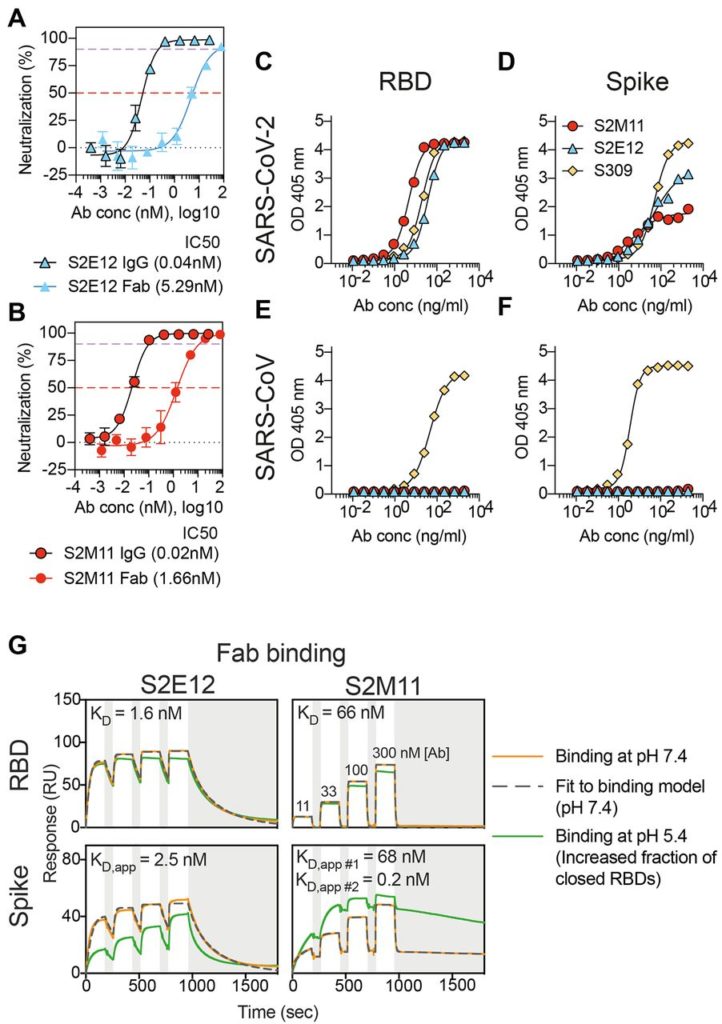
Per identificare mAbs altamente potenti provocati dall’infezione da SARS-CoV-2, abbiamo ordinato le cellule B della memoria da due individui che si stanno riprendendo da una grave malattia COVID-19, utilizzando il trimero di ectodominio SARS-CoV-2 S di prefusione biotinilata come esca. Due mAb, S2E12 e S2M11, si sono distinti per la loro elevata potenza di neutralizzazione contro il virus SARS-CoV-2 autentico e due diversi virus pseudotipati SARS-CoV-2 S (utilizzando il virus della leucemia murina (MLV) o il virus della stomatite vescicolare (VSV) spine dorsali). In un saggio che misura l’inibizione dell’ingresso autentico di SARS-CoV-2 (SARS-CoV-2-Nluc ( 35 )), abbiamo determinato concentrazioni inibitorie metà massime (IC 50 ) di 3-6 ng / ml (20-40 pM ) ( Fig.1, A e B ). Abbiamo determinato IC 50valori di 1,9-2,5 ng / ml per SARS-CoV-2 S-VSV (fig. S1A) e 10,3-30,4 ng / ml per SARS-CoV-2 S-MLV (fig. S1B). In un autentico test di neutralizzazione della riduzione del fuoco SARS-CoV-2 che misura l’inibizione dell’ingresso e della diffusione del virus ( 36 ), i valori di IC 50 erano 1,2-6,6 ng / ml (fig. S1C). L’eccezionale potenza di questi mAb è stata ulteriormente dimostrata dalle concentrazioni necessarie per inibire il 90% dell’entrata virale autentica di SARS-CoV-2-Nluc (IC 90 ), che abbiamo determinato come 26,4 ± 7,8 ng / ml e 12,7 ± 3,1 ng / ml rispettivamente per S2E12 e S2M11 ( Fig. 1, A e B ). La maggiore potenza di neutralizzazione delle IgG rispetto a Fab osservata per ciascun mAb ha suggerito che le distinte affinità di legame e / o il legame bivalente contribuiscono alla potenza (Fig.1, A e B ). La catena pesante S2E12 utilizza i geni VH1-58 * 01, D2-15 * 01 e JH3 * 02 mentre S2M11 deriva dai geni VH1-2 * 02, D3-3 * 01 e JH4 * 02. L’identità germinale della sequenza nucleotidica del gene variabile della catena pesante è del 96,53% per S2M11 e del 97,6% per S2E12, mostrando un basso livello di ipermutazione somatica per questi due mAb.
La sindrome respiratoria acuta grave Coronavirus 2 (SARS-CoV-2) è emersa alla fine del 2019 ed è stata sequenziata entro gennaio 2020 ( 1 , 2 ). Sebbene l’ospite del serbatoio responsabile della ricaduta nella popolazione umana rimanga incerto, SARS-CoV-2 sembra aver avuto origine in pipistrelli da cui sono stati identificati virus e sequenze virali strettamente correlati ( 1 , 3 ). SARS-CoV-2 appartiene al sottogenere del sarbecovirus ed è strettamente correlato al SARS-CoV, responsabile di un’epidemia nel 2002-2003 che ha provocato 8.098 casi e 774 decessi in tutto il mondo ( 4 , 5). La mancanza di immunità preesistente alla SARS-CoV-2 a causa della sua divergenza dai quattro coronavirus endemici circolanti e la sua elevata trasmissibilità da uomo a uomo ha portato alla pandemia COVID-19 in corso che ha già causato oltre 29 milioni infezioni e oltre 922.000 decessi a metà settembre 2020.
L’infezione da SARS-CoV-2 inizia con l’attaccamento della glicoproteina virale transmembrana spike (S) tramite un motivo di legame del recettore (RBM) all’enzima di conversione dell’angiotensina 2 (ACE2), che porta alla fusione della membrana e all’ingresso nelle cellule ospiti ( 6 – 13 ). Come per tutti i coronavirus, SARS-CoV-2 S è l’obiettivo principale degli anticorpi neutralizzanti (Abs) e un fulcro della progettazione del vaccino e degli sforzi di targeting terapeutico ( 14 ). Anche se i programmi di sviluppo di vaccini sono una corsia preferenziale ( 15 – 20), la produzione e l’amministrazione su larga scala per una popolazione sufficientemente ampia da ottenere la protezione della comunità richiederanno probabilmente molti mesi. I farmaci antivirali profilattici e / o terapeutici potrebbero colmare il divario prima che vaccini sicuri ed efficienti diventino ampiamente disponibili e continueranno ad avere utilità in individui non vaccinati o in coloro che rispondono male alla vaccinazione.
Abbiamo recentemente descritto un Ab monoclonale (mAb), isolato dalle cellule B della memoria di un sopravvissuto alla SARS ottenuto 10 anni dopo il recupero, che neutralizza SARS-CoV-2 e SARS-CoV attraverso il riconoscimento del dominio di legame del recettore S (RBD) ma senza bloccare l’attacco ACE2 ( 21 ). Una versione ottimizzata di S309 è attualmente in fase di valutazione in studi clinici di fase 2/3. L’isolamento di molti altri RBD mirati neutralizzando Abs da COVID-19 pazienti convalescenti ( 22 – 28 ) e la dimostrazione che essi forniscono nella protezione vivo contro la SARS-CoV-2 sfida nei piccoli animali e primati non umani ( 25 , 29 – 31) hanno dimostrato che l’RBD è l’obiettivo principale della neutralizzazione degli addominali in caso di infezione da CoV naturale. La valutazione clinica di Abs terapeutico interferire direttamente con il legame ACE2 sono in corso ( il 30 – 34 ). Gli mAb con una potenza di neutralizzazione eccezionalmente elevata, insieme a meccanismi d’azione distinti e complementari rispetto agli mAb esistenti, possono consentire la formulazione di cocktail mAb con maggiore efficacia per controllare la diffusione del virus e prevenire la resistenza. Qui, abbiamo valutato la possibilità di combinare due Abs neutralizzanti ultrapotenti che abbiamo scoperto, ovvero S2E12 e S2M11, che sfruttano diversi meccanismi di azione.
Risultati
 Isolamento di Abs neutralizzante SARS-CoV-2 ultrapotente
Isolamento di Abs neutralizzante SARS-CoV-2 ultrapotente
Per identificare mAbs altamente potenti provocati dall’infezione da SARS-CoV-2, abbiamo ordinato le cellule B della memoria da due individui che si stanno riprendendo da una grave malattia COVID-19, utilizzando il trimero di ectodominio SARS-CoV-2 S di prefusione biotinilata come esca. Due mAb, S2E12 e S2M11, si sono distinti per la loro elevata potenza di neutralizzazione contro il virus SARS-CoV-2 autentico e due diversi virus pseudotipati SARS-CoV-2 S (utilizzando il virus della leucemia murina (MLV) o il virus della stomatite vescicolare (VSV) spine dorsali). In un saggio che misura l’inibizione dell’ingresso autentico di SARS-CoV-2 (SARS-CoV-2-Nluc ( 35 )), abbiamo determinato concentrazioni inibitorie metà massime (IC 50 ) di 3-6 ng / ml (20-40 pM ) ( Fig.1, A e B ). Abbiamo determinato IC 50valori di 1,9-2,5 ng / ml per SARS-CoV-2 S-VSV (fig. S1A) e 10,3-30,4 ng / ml per SARS-CoV-2 S-MLV (fig. S1B). In un autentico test di neutralizzazione della riduzione del fuoco SARS-CoV-2 che misura l’inibizione dell’ingresso e della diffusione del virus ( 36 ), i valori di IC 50 erano 1,2-6,6 ng / ml (fig. S1C). L’eccezionale potenza di questi mAb è stata ulteriormente dimostrata dalle concentrazioni necessarie per inibire il 90% dell’entrata virale autentica di SARS-CoV-2-Nluc (IC 90 ), che abbiamo determinato come 26,4 ± 7,8 ng / ml e 12,7 ± 3,1 ng / ml rispettivamente per S2E12 e S2M11 ( Fig. 1, A e B ). La maggiore potenza di neutralizzazione delle IgG rispetto a Fab osservata per ciascun mAb ha suggerito che le distinte affinità di legame e / o il legame bivalente contribuiscono alla potenza (Fig.1, A e B ). La catena pesante S2E12 utilizza i geni VH1-58 * 01, D2-15 * 01 e JH3 * 02 mentre S2M11 deriva dai geni VH1-2 * 02, D3-3 * 01 e JH4 * 02. L’identità germinale della sequenza nucleotidica del gene variabile della catena pesante è del 96,53% per S2M11 e del 97,6% per S2E12, mostrando un basso livello di ipermutazione somatica per questi due mAb.
Sia S2E12 che S2M11 sono legati al SARS-CoV-2 RBD e al trimero S ectodominio stabilizzato alla prefusione ( 6 ) ma non al SARS-CoV RBD o S ( 37 ) da ELISA ( Fig.1, da C a F). Utilizzando la risonanza plasmonica di superficie (SPR) e la citometria a flusso, abbiamo inoltre osservato che S2E12 e S2M11 competono per il legame al SARS-CoV-2 RBD o al SARS-CoV-2 S, presentato come trimero dell’ectodominio S stabilizzato in prefusione o come S a tutta lunghezza espressa sulla superficie delle cellule ExpiCHO (fig. S2, A e B). Quando è stato aggiunto per primo, S2M11 ha gareggiato in modo dipendente dalla concentrazione con il sarbecovirus neutralizzando S309 mAb per legarsi a SARS-CoV-2 S, mentre potrebbe legarsi con una concorrenza minima quando aggiunto dopo S309 (fig. S2B). Mentre il Fab S2E12 (o IgG) si lega a SARS-CoV-2 S e RBD in modo simile, l’affinità di legame del Fab S2M11 (o IgG) per il trimero S è stata migliorata rispetto al RBD SARS-CoV-2 isolato ( Fig. 1Ge la fig. S2C). In particolare, la cinetica di legame di S2M11 a SARS-CoV-2 S era bifasica, inclusa una prima fase con cinetica di legame e affinità identiche misurate per il legame all’RBD isolato, e una seconda fase con un off-rate molto più lento e quindi una maggiore affinità. Abbiamo osservato che il legame di S2M11 Fab e IgG a S era aumentato a pH 5,4, una condizione che favorisce la conformazione del trimero chiuso, rispetto a pH 7,4 ( 38 ) ( Fig. 1G , Fig. S2C e tabella S1). Al contrario, il legame di S2E12 Fab a S era diminuito a pH 5,4 (e moderatamente ridotto per S2E12 IgG), probabilmente a causa dell’aumentato numero di trimeri S con RBD chiusi ( Fig. 1G ; fig. S2, A e C; e tabella S1).
Collettivamente, questi risultati indicano che S2E12 e S2M11 hanno come target epitopi RBD SARS-CoV-2 sovrapposti o parzialmente sovrapposti. La scoperta che S2M11 interagisce preferenzialmente con il trimero S rispetto al RBD suggerisce che questo mAb potrebbe legarsi a un epitopo quaternario esposto solo nel contesto di una prefusione chiusa nativa S. Infine, il legame potenziato di S2E12 a SARS-CoV-2 S in condizioni che favoriscono l’apertura RBD (pH 7.4) indica che questo mAb potrebbe riconoscere un epitopo criptico non esposto nel trimero S chiuso.
S2E12 neutralizza potentemente SARS-CoV-2 prendendo di mira l’RBM
Per comprendere il meccanismo della potente neutralizzazione mediata da S2E12 di SARS-CoV-2, abbiamo caratterizzato un complesso tra il trimero dell’ectodominio SARS-CoV-2 S e il frammento Fab S2E12 mediante microscopia crioelettronica (cryoEM). La classificazione 3D dei dati ha mostrato la presenza di trimeri S con uno, due o tre Fab legati ad RBD aperti per i quali abbiamo determinato strutture con risoluzione di 3.5 Å, 3.3Å e 3.3 Å, rispettivamente (Fig.2 , A e B; Figura. S3, da A a G; e tabella S2). Successivamente abbiamo utilizzato il raffinamento locale per ottenere una mappa 3,7 Å della regione corrispondente ai domini variabili S2E12 e RBD, che ha notevolmente migliorato la risoluzione locale a causa della dinamica conformazionale rispetto al resto del trimero S, e l’abbiamo usata insieme a un cristallo 1,4Å struttura della S2E12 Fab per costruire un modello (fig. S3, da D a G, e tabelle S2 e S3).
 Fig. 2 L’mAb neutralizzante S2E12 riconosce l’RBM SARS-CoV-2.
Fig. 2 L’mAb neutralizzante S2E12 riconosce l’RBM SARS-CoV-2.
( A e B ) Struttura CryoEM del trimero di ectodominio SARS-CoV-2 S a prefusione con tre frammenti Fab S2E12 legati a tre RBD aperti visti lungo due orientamenti ortogonali. ( C ) Il paratopo concavo S2E12 riconosce la punta convessa RBM. ( D ) Vista ravvicinata che mostra le interazioni selezionate formate tra S2E12 e SARS-CoV-2 RBD. Nei pannelli AD, ogni protomero SARS-CoV-2 S è colorato distintamente (ciano, rosa e oro) mentre i domini variabili della catena leggera e pesante S2E12 sono colorati rispettivamente di magenta e viola. I glicani legati all’N sono rappresentati come sfere blu nei pannelli AC.
S2E12 riconosce un epitopo RBD che si sovrappone al RBM (cioè, il sito di legame del recettore ACE2) che è parzialmente sepolto all’interfaccia tra i protomeri nel trimero S chiuso ( Fig. 2, da A a D , e fig. S4, A e B) . Di conseguenza, S2E12 può interagire solo con RBD aperti, come nel caso di ACE2 e di diversi mAb neutralizzanti precedentemente descritti tra cui S2H14 ( 22 , 25 , 28 ). Il paratopo concavo S2E12 riconosce la punta convessa RBM attraverso interazioni elettrostatiche e di van der Waals (Fig.2 , C e D). In particolare, S2E12 utilizza le regioni determinanti complementari della catena pesante (CDR) 1-3 e le catene leggere CDR1 e CDR3, che rappresentano rispettivamente 2/3 e 1/3 della superficie sepolta del paratopo, per riconoscere i residui 455-458 e 473- 493 del SARS-CoV-2 RBD ( Fig.2, C e D ). Praticamente tutti i contatti S2E12 con il RBD sono mediati da residui codificati della linea germinale con solo 1 residuo mutato di catena pesante su 5 (G109) e 1 su 4 di catena leggera (G94) che contribuiscono al paratopo. I dati strutturali spiegano che S2E12 si lega efficacemente sia al RBD che al trimero S di prefusione ( Fig. 1G ) e neutralizza potentemente SARS-CoV-2 ( Fig. 1, A e Be la fig. S1, A e C): (i) S2E12 riconosce un epitopo 3D terziario, cioè un epitopo che è completamente contenuto in un protomero S; (ii) ~ 50% dei trimeri S ospita naturalmente un RBD aperto sulla superficie virale o in trimeri S ectodominio espressi in modo ricombinante, come osservato dalla tomografia crioelettronica e dal crioEM a singola particella, rispettivamente ( 6 , 39 ) e (iii) legame S2E12 sposta l’equilibrio conformazionale RBD verso trimeri S aperti, come descritto in precedenza per mAbs mirati RBM ( 22 , 28 , 37 ).
S2M11 blocca il trimero SARS-CoV-2 S nello stato chiuso legandosi a un epitopo quaternario
Abbiamo effettuato analisi cryoEM di S2M11 in complesso con SARS-CoV-2 S per chiarire le basi molecolari del suo riconoscimento preferenziale del trimero S rispetto al RBD e il suo meccanismo di neutralizzazione. La classificazione 3D dei dati cryoEM ha rivelato la presenza esclusiva di trimeri S adottando una conformazione chiusa, che ci ha permesso di determinare una struttura 2.6Å di SARS-CoV-2 S legati a tre frammenti S2M11 Fab (Fig.3 , A e B ; fig. . S5, da A a F e tabella S2). S2M11 riconosce un epitopo quaternario attraverso interazioni elettrostatiche e complementarità di forma, comprendente regioni distinte di due RBD adiacenti all’interno di un trimero S (Fig.3 , C e D). In particolare, S2M11 CDRH1, CDRH2 e la regione 3 della struttura della catena pesante (FR3) sono ancorate nella fessura dell’RBM (seppellendo una superficie di ~ 400Å 2 ) mentre CDRH3 copre l’interfaccia tra l’RBM e le eliche 339-343, 367-374 pure come residuo 436 di un RBD adiacente appartenente al protomero adiacente (cioè, seppellendo una superficie totale di ~ 500 Å 2 ) ( Fig. 3, C e F ). Sebbene la maggior parte delle interazioni siano mediate dalla catena pesante S2M11, CDRL2 interagisce con i residui 440-441 e CDRL1 forma contatti chiave con il glicano nella posizione N343, che è ruotato di ~ 45 ° rispetto all’orientamento che adotta nella struttura S associata a S309 ( 21 ), entrambi i gruppi di interazioni che si verificano con il vicino RBD (epitopo quaternario) ( Fig.3, C e Fe la fig. S5G). Tre degli otto residui della catena pesante S2M11 che sono mutati rispetto alla linea germinale contribuiscono al riconoscimento degli epitopi (Ile54, Thr77 e Phe102) mentre nessuno dei due residui mutati della catena leggera partecipa al legame RBD.
S2E12 riconosce un epitopo RBD che si sovrappone al RBM (cioè, il sito di legame del recettore ACE2) che è parzialmente sepolto all’interfaccia tra i protomeri nel trimero S chiuso ( Fig. 2, da A a D , e fig. S4, A e B) . Di conseguenza, S2E12 può interagire solo con RBD aperti, come nel caso di ACE2 e di diversi mAb neutralizzanti precedentemente descritti tra cui S2H14 ( 22 , 25 , 28 ). Il paratopo concavo S2E12 riconosce la punta convessa RBM attraverso interazioni elettrostatiche e di van der Waals (Fig.2 , C e D). In particolare, S2E12 utilizza le regioni determinanti complementari della catena pesante (CDR) 1-3 e le catene leggere CDR1 e CDR3, che rappresentano rispettivamente 2/3 e 1/3 della superficie sepolta del paratopo, per riconoscere i residui 455-458 e 473- 493 del SARS-CoV-2 RBD ( Fig.2, C e D ). Praticamente tutti i contatti S2E12 con il RBD sono mediati da residui codificati della linea germinale con solo 1 residuo mutato di catena pesante su 5 (G109) e 1 su 4 di catena leggera (G94) che contribuiscono al paratopo. I dati strutturali spiegano che S2E12 si lega efficacemente sia al RBD che al trimero S di prefusione ( Fig. 1G ) e neutralizza potentemente SARS-CoV-2 ( Fig. 1, A e Be la fig. S1, A e C): (i) S2E12 riconosce un epitopo 3D terziario, cioè un epitopo che è completamente contenuto in un protomero S; (ii) ~ 50% dei trimeri S ospita naturalmente un RBD aperto sulla superficie virale o in trimeri S ectodominio espressi in modo ricombinante, come osservato dalla tomografia crioelettronica e dal crioEM a singola particella, rispettivamente ( 6 , 39 ) e (iii) legame S2E12 sposta l’equilibrio conformazionale RBD verso trimeri S aperti, come descritto in precedenza per mAbs mirati RBM ( 22 , 28 , 37 ).
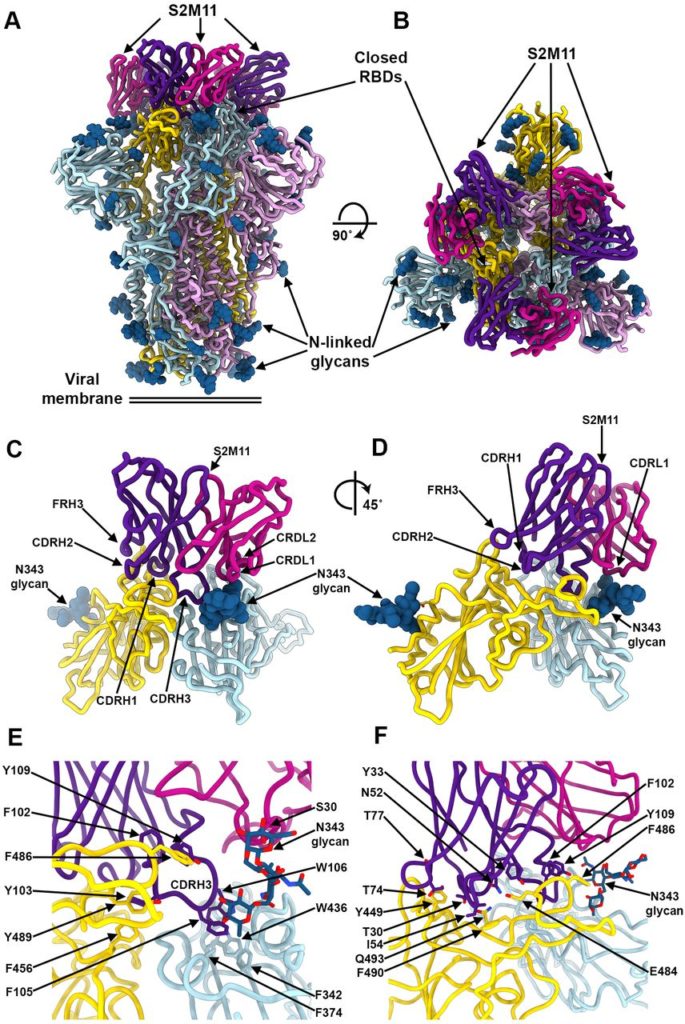 Fig. 3 L’mAb neutralizzante S2M11 riconosce un epitopo quaternario che si estende su due RBD e stabilizza S nello stato chiuso.
Fig. 3 L’mAb neutralizzante S2M11 riconosce un epitopo quaternario che si estende su due RBD e stabilizza S nello stato chiuso.
( A e B ) Struttura CryoEM del trimero di ectodominio SARS-CoV-2 S di prefusione legato a tre frammenti Fab S2M11 visti lungo due orientamenti ortogonali. ( C e D ) La posa di legame S2M11, che coinvolge un epitopo quaternario che abbraccia due RBD vicini. ( E e F ) Viste ravvicinate che mostrano interazioni selezionate formate tra S2M11 e RBD SARS-CoV-2. Nei pannelli AF, ogni protomero SARS-CoV-2 S è colorato distintamente (ciano, rosa e oro) mentre i domini variabili della catena leggera e pesante S2M11 sono colorati rispettivamente di magenta e viola. I glicani legati all’N sono rappresentati come sfere blu nei pannelli AD e come bastoncini nei pannelli EF. FR: framework.
L’osservazione che tutte le immagini di particelle corrispondono a trimeri S chiusi quando legati a S2M11 contrasta con la nostra precedente scoperta di ~ 50% / 50% di trimeri chiusi o con un RBD aperto in assenza di mAb legato ( 6 ) o in complesso con S309 ( 21 ) o S2H13 ( 28 ), che non selezionano per alcuna specifica conformazione RBD. Sulla base di questi dati, concludiamo che S2M11 stabilizza la conformazione chiusa del trimero S interagendo con un epitopo composito che include due RBD vicini (da due protomeri distinti) che sono vicini l’uno all’altro nello stato chiuso ma si allargano all’apertura RBD ( 6 ) (fig. S4, C e D). Questi risultati spiegano anche la maggiore affinità di legame di S2M11 per S rispetto a RBD ( Fig. 1G), poiché solo il trimero S consente il legame all’epitopo quaternario che seppellisce una superficie del paratopo maggiore del ~ 60% rispetto al legame con l’RBM isolato (Fig.3 , da A a F ). Interpretiamo quindi il legame bifasico come S2M11 che interagisce con un epitopo terziario presente in RBD aperti (off-rate veloce), in base alla cinetica e affinità identiche misurate rispetto a RBD isolato, e S2M11 che riconosce il suo epitopo quaternario completo (off-rate lento) .
S2M11 e S2E12 inibiscono l’attaccamento di SARS-CoV-2 ad ACE2 e attivano le funzioni effettrici mediate da Fc
I dati strutturali indicano che sia S2E12 che S2M11 competerebbero con l’attaccamento di ACE2 all’RBD poiché riconoscono epitopi che si sovrappongono con l’RBM ( Fig.4, A e B ). Inoltre, la stabilizzazione indotta da S2M11 di SARS-CoV-2 S nello stato conformazionale chiuso produce trimeri S con RBM mascherati che sono incompetenti per l’impegno del recettore, come mostrato in precedenza per un costrutto S ingegnerizzato stabilizzato in modo covalente nello stato chiuso ( 40 ). Quindi, sia S2E12 che S2M11 hanno bloccato il legame di SARS-CoV-2 S o RBD con ACE2 ricombinante umano immobilizzato misurato mediante interferometria biostrato ( Fig. 4, C e D ). Inoltre, sia S2E12 che S2M11 hanno inibito il legame di ACE2 a SARS-CoV-2 S espresso sulla superficie delle cellule CHO ( Fig. 4E), convalidando questo meccanismo di neutralizzazione utilizzando trimeri S nativi a lunghezza intera. L’efficienza comparabile di S2E12 e S2M11 nel bloccare l’attaccamento di S ad ACE2 è correlata alle loro simili potenze di neutralizzazione.
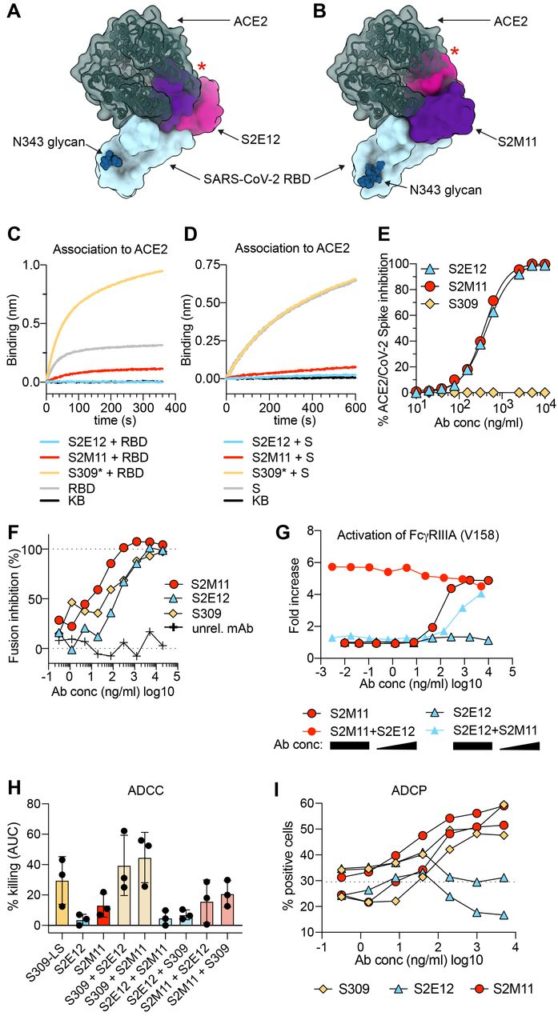 Fig. 4 S2E12 e S2M11 impediscono l’attaccamento di SARS-CoV-2 S ad ACE2, inibiscono la fusione della membrana e S2M11 innesca le funzioni effettrici.
Fig. 4 S2E12 e S2M11 impediscono l’attaccamento di SARS-CoV-2 S ad ACE2, inibiscono la fusione della membrana e S2M11 innesca le funzioni effettrici.
( A ) S2E12 (viola / rosa) e ACE2 (verde scuro) si legano a siti di legame sovrapposti sul SARS-CoV-2 RBD (blu). ( B ) S2M11 (viola / rosa) e ACE2 (verde scuro) si legano a siti di legame sovrapposti sul SARS-CoV-2 RBD (blu). Le stelle rosse indicano scontri sterici. ( C e D ) Legame del trimero SARS-CoV-2 RBD (C) o S ectodominio (D) da solo (grigio) o precomposto con S2M11 (rosso), S2E12 (blu) o S309 * (giallo) mAb al Ectodominio ACE2 immobilizzato sulla superficie dei biosensori analizzati mediante interferometria biostrato. S309 * è una versione ottimizzata del genitore S309 mAb ( 21 ). KB: buffer cinetico (controllo negativo). ( E) Legame di concentrazioni variabili di mAbs S2E12 (blu), S2M11 (rosso) o S309 (giallo) a S a lunghezza intera espressa sulla superficie delle cellule CHO in presenza di 20 μg / mL dell’ectodominio ACE2 analizzato mediante citometria a flusso ( una misurazione per condizione). ( F ) Test di inibizione della fusione cellula-cellula con cellule Vero E6 trasfettate con SARS-CoV-2 S e incubate con concentrazioni variabili di mAb S2E12 (blu), S2M11 (rosso) o S309 (giallo) e un mAb di controllo. I valori sono normalizzati alla percentuale di fusione senza mAb e alla percentuale di fusione di cellule non trasfettate. ( G) Segnalazione FcγRIIIa (variante V158 ad alta affinità) indotta da singoli mAb o cocktail mAb. Per i cocktail mAb, la concentrazione del mAb costante era 5 μg / ml. La concentrazione di mAb diluito è indicata sull’asse x. ( H ) ADCC che utilizza cellule NK primarie come effettori e cellule CHO che esprimono S SARS-CoV-2 come bersagli. L’entità dell’uccisione mediata dalle cellule NK è espressa come l’area sotto la curva (AUC) per ciascun mAb utilizzato a concentrazioni comprese tra 0,1 ng / ml e 20 μg / ml. Per i cocktail mAb, il primo mAb elencato è stato mantenuto costante a 5 μg / ml. Ogni simbolo rappresenta un donatore, i dati vengono combinati da due esperimenti individuali. Vedi fig. S6E per le curve di un donatore rappresentativo. ( Io) ADCP che utilizza PBMC come fonte di cellule fagocitiche (monociti) e cellule CHO che esprimono S fluorescenti marcate PKH67 come cellule bersaglio. L’asse y indica la% di monociti doppiamente positivi per il marker anti-CD14 (monociti) e PKH67. La linea tratteggiata indica il segnale rilevato in presenza di cellule bersaglio ed effettrici ma senza mAb (linea di base). Ogni riga indica i dati per un donatore PBMC. I simboli sono mezzi di duplicati. I dati provengono da un esperimento. Ab conc: concentrazione mAb
Per indagare ulteriormente il meccanismo di inibizione della SARS-CoV-2 da parte di S2E12 e S2M11, abbiamo eseguito un test di fusione cellula-cellula utilizzando cellule VeroE6 (che esprimono endogenamente ACE2 sulla loro superficie) transfettate transitoriamente con SARS-CoV-2 S di tipo selvatico a lunghezza intera Sebbene S2E12 e S2M11 si leghino e stabilizzino differenti conformazioni della proteina S, entrambi gli mAb hanno bloccato efficacemente la formazione di sincizi ( Fig. 4F ), che risulta dalla fusione della membrana mediata da S. L’assenza di formazione di sincizi è probabilmente spiegata dalla rottura mediata da S2E12 o S2M11 del legame ACE2 insieme all’inibizione della fusione di membrana indotta da S2M11 attraverso intrappolamento conformazionale di SARS-CoV-2 S nello stato chiuso.
La citotossicità cellulare dipendente da anticorpi (ADCC) mediata da cellule natural killer o la fagocitosi cellulare dipendente da ab (ADCP) mediata da macrofagi o monociti sono funzioni effettrici mediate da Fc che possono contribuire alla protezione facilitando l’eliminazione del virus e supportando le risposte immunitarie in vivo , indipendentemente dalla neutralizzazione diretta ( 41). Come prerequisito per la comparsa dell’ADCC, abbiamo convalidato che le cellule infette esprimono SARS-CoV-2 S sulla loro superficie (fig. S6, A e B). Per valutare la capacità di S2M11 e S2E12 di sfruttare ADCC e ADCP, abbiamo testato se questi mAbs (backbone IgG1) potessero indurre la segnalazione mediata da FcγRIIa e FcγRIIIa utilizzando un saggio reporter della luciferasi. S2M11 ha promosso un segnale efficiente, dose-dipendente mediato da FcγRIIIa (ma non mediato da FcγRIIa), in particolare per la variante ad alta affinità (V158) del recettore Fc, a livelli paragonabili all’mAb cross-reattivo S309 ( Fig. 4G e fig. . S6, C e D) ( 21). Al contrario, S2E12 ha attivato la segnalazione mediata da FcγRIIa (ma non mediata da FcγRIIIa), probabilmente come risultato del distinto orientamento dell’mAb rispetto alla membrana delle cellule effettrici rispetto a S2M11 e S309 ( Fig. 4G e fig. S6C ). Di conseguenza, S2M11 ma non S2E12 hanno mostrato attività ADCC dipendente da FcγRIIIa ( Fig. 4H e fig. S6E) e attività ADCP ( Fig. 4I ). Poiché abbiamo osservato un’efficace attivazione delle funzioni effettrici mescolando S2M11 con S2E12 o S309 ( Fig. 4, G e H e fig. S6E), proponiamo che i cocktail di questi mAbs possano sfruttare meccanismi protettivi aggiuntivi in vivo oltre all’inibizione dell’ingresso virale.
Formulazione di cocktail Ab neutralizzanti ultrapotenti contro SARS-CoV-2
Gli sforzi di sorveglianza hanno portato all’identificazione di una serie di mutanti S tra gli isolati di SARS-CoV-2 circolanti. È stato dimostrato che diverse mutazioni RBD presenti in natura abrogano le interazioni con mAb noti e riducono il legame del siero immunitario, sollevando la preoccupazione che mutanti di fuga dalla neutralizzazione virale possano emergere o essere selezionati sotto pressione da trattamenti antivirali basati su mAb ( 42). Per indagare se la neutralizzazione mediata da S2E12 e S2M11 possa essere influenzata dal polimorfismo SARS-CoV-2, abbiamo testato il legame di entrambi gli mAb alle varianti della proteina 29 S (corrispondenti alle mutazioni rilevate negli isolati circolanti di SARS-CoV-2) espresse in superficie di cellule CHO. Le varianti Y449N, E484K / Q, F490L e S494P RBD hanno portato a una diminuzione del legame di S2M11 con S mentre nessuno dei mutanti ha testato le interazioni influenzate con S2E12, sebbene molti di loro si trovino nell’epitopo di quest’ultimo mAb (tabella S4). L’impatto di queste sostituzioni sul legame S2M11 è spiegato dai dati strutturali che mostrano che le catene laterali SARS-CoV-2 S Y449 ed E484 sono legate a idrogeno all’ammide dorsale della catena pesante S2M11 F29 e alle catene laterali N52 / S55, rispettivamente, ei residui di F490 e S494 vengono sepolti all’interfaccia con S2M11.I test di ingresso del virus pseudotipato SARS-CoV-2 S-VSV con varianti S selezionate hanno confermato questi risultati e hanno mostrato che le singole sostituzioni Y449N, E484K / Q, F490L / S e S494P hanno abrogato la neutralizzazione mediata da S2M11 mentre la variante L455F ha ridotto la potenza di neutralizzazione di un ordine di grandezza (fig. S7, A, C ed E). S2E12 ha neutralizzato in modo efficiente tutte le varianti testate eccetto G476S che ha mostrato un ordine di grandezza diminuito della potenza (fig. S7, B, D e F). In accordo con i dati di scansione mutazionale profonda (S2E12 ha neutralizzato in modo efficiente tutte le varianti testate eccetto G476S che ha mostrato un ordine di grandezza diminuito di potenza (fig. S7, B, D e F). In accordo con i dati di scansione mutazionale profonda (S2E12 ha neutralizzato in modo efficiente tutte le varianti testate eccetto G476S che ha mostrato una potenza ridotta in ordine di grandezza (fig. S7, B, D e F). In accordo con i dati di scansione mutazionale profonda (43 ), abbiamo scoperto che la variante Y449N era compromessa nella sua capacità di legare ACE2 (fig. S8) che dovrebbe ridurre l’idoneità virale, probabilmente spiegando che questa mutazione è stata segnalata fino ad oggi solo in uno dei 90.287 SARS-CoV completi -2 sequenze genomiche. Sebbene rare, le mutazioni G476S, E484K / Q, S494P e F490L / S sono state rilevate in 20, 10/17, 15 e 5/8 isolati virali e in teoria potrebbero essere selezionate sotto la pressione selettiva di S2E12 o S2M11. Complessivamente, sono state riportate quindici varianti di SARS-CoV-2 S con una singola sostituzione amminoacidica all’interno dell’epitopo S2M11, con una prevalenza inferiore allo 0,1% a settembre 2020 (fig. S7G).
Per aggirare il rischio di comparsa o selezione di mutanti di fuga dalla neutralizzazione, abbiamo valutato se S2M11, S2E12 e S309 potessero essere combinati in cocktail mAb a due componenti in base ai loro meccanismi d’azione complementari. I saggi di ingresso del virus pseudotipato SARS-CoV-2 S-VSV hanno mostrato che i cocktail mAb neutralizzavano potentemente le varianti Y449N, S494P e G476S e superavano il fenotipo di fuga di neutralizzazione osservato con singoli mAb (fig. S7, da H a J). Una matrice di concentrazione di S2E12 e S2M11 ha rivelato i loro effetti di neutralizzazione additiva senza antagonismo, nonostante il fatto che entrambi gli Abs competano per il legame con l’RBM (fig. S9, da A a C). Inoltre, la combinazione di S309 con S2E12, che non competono per l’associazione a S, e S309 e S2M11, che competono parzialmente (cioè per il fissaggio al trimer S chiuso),ha anche prodotto effetti di neutralizzazione additiva (fig. S9, da D a F), suggerendo che i cocktail mAb a due (o tre) componenti sono una strategia terapeutica promettente per prevenire l’emergenza o la selezione di mutanti virali che sfuggono alla terapia con mAb.
S2M11 e S2E12 proteggono i criceti dalla sfida SARS-CoV-2
Per valutare l’efficacia protettiva di S2E12 e S2M11 contro la sfida SARS-CoV-2 in vivo, sono stati testati mAb o un cocktail di entrambi gli mAb in un modello di criceto siriano ( 44 ). Gli mAb sono stati progettati con regioni costanti della catena pesante e leggera dell’IgG2 di criceto siriano per consentire l’attivazione ottimale delle funzioni effettrici dipendenti da Fc. Gli mAb sono stati somministrati tramite iniezione intraperitoneale 48 ore prima della stimolazione intranasale con 2 × 10 6 TCID 50 di SARS-CoV-2. Quattro giorni dopo, i polmoni sono stati raccolti per la quantificazione dell’RNA virale e del virus infettivo. Il solo mAb o i cocktail con 0,5 mg / kg o 1 mg / kg di mAb totale hanno ridotto la quantità di RNA virale rilevata nei polmoni da 2 a 5 ordini di grandezza rispetto ai criceti che hanno ricevuto un mAb di controllo ( Fig. 5A). Le quantità di RNA virale rilevate al giorno 4 erano inversamente correlate con la concentrazione di mAb sierica misurata al momento dell’infezione (Spearman R -0,574, p = 0,0052) ( Fig. 5B ). La somministrazione profilattica di questi mAb a tutte le dosi testate ha completamente abrogato la replicazione virale nei polmoni, ad eccezione di un singolo animale che ha ricevuto il cocktail a basso dosaggio ed è stato parzialmente protetto ( Fig. 5C ). Questi dati mostrano una notevole efficacia protettiva di entrambi gli mAb a basse dosi, singolarmente o come cocktail, in linea con la loro neutralizzazione in vitro ultrapotente.
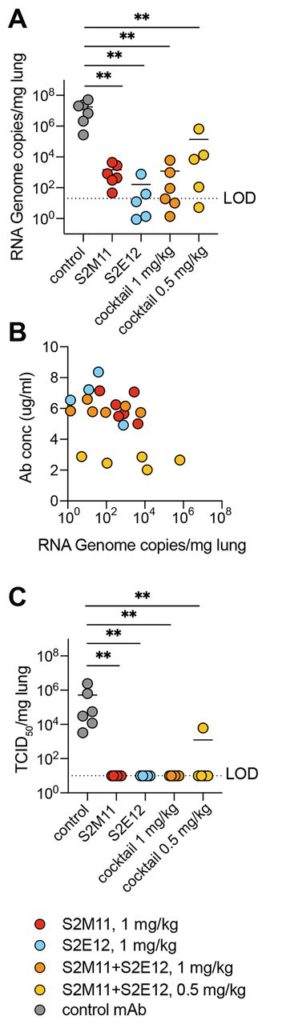 Fig. 5 S2E12, S2M11 o cocktail dei due mAb forniscono una solida protezione in vivo contro la sfida SARS-CoV-2.
Fig. 5 S2E12, S2M11 o cocktail dei due mAb forniscono una solida protezione in vivo contro la sfida SARS-CoV-2.
I criceti siriani sono stati iniettati con la quantità indicata di mAb 48 ore prima della sfida intra-nasale con SARS-CoV-2. ( A ) Quantificazione dell’RNA virale nei polmoni 4 giorni dopo l’infezione. ( B ) La concentrazione di mAbs misurata nel siero prima dell’infezione (giorno 0) è inversamente correlata alla carica di RNA virale nel polmone 4 giorni dopo l’infezione. ( C ) Quantificazione del virus replicante negli omogenati polmonari raccolti 4 giorni dopo l’infezione utilizzando un dosaggio TCID 50 . Per i cocktail mAb, è indicata la dose totale di una miscela equimolare di entrambi gli mAb
Discussione
S2M11 e S2E12 sono stati identificati tra quasi 800 addominali selezionati isolati da 12 individui che si sono ripresi da COVID-19. L’ultrapotenza e l’epitopo quaternario di S2M11 sembrano essere rari rispetto ai più canonici Abs neutralizzanti specifici per RBM, poiché quest’ultimo tipo di mAbs era presente in ogni donatore che abbiamo analizzato. Un mAb che riconosceva la conformazione S chiusa (mAb 2-43) è stato precedentemente identificato e la mappatura a bassa risoluzione del suo sito di legame ha suggerito che potrebbe interagire con un epitopo quaternario che appare distinto da quello di S2M11 ( 45 ). Due rapporti recenti descrivono l’identificazione di un mAb e di un nanobody che prende di mira gli epitopi quaternari, che abbraccia due RBD vicini, che sono presenti nel trimero S chiuso. Nb6 è stato identificato da una libreria naïve di nanobody, affinità maturata e trimerizzata per ottenere un CI50 di 160 pM, tuttavia senza la capacità di esercitare le funzioni effettive ( 46 ). C144 è stato isolato da un campione di siero convalescente COVID-19, utilizza i geni VH3-53 e VL2-14, ospita un CDRH3 lungo 25 residui e neutralizza potentemente SARS-CoV-2 ( 47 ). Simile a S2M11, Nb6 (insieme ai suoi derivati ingegnerizzati) e C144 usano CDR (H) 3 per collegare due RBD vicini e stabilizzare SARS-CoV-2 S nello stato chiuso. Una lunga CDRH3 di 15 o più residui amminoacidici era una caratteristica comune degli mAb di tipo C144 ( 47 ). Contrariamente al CDRH3 lungo a 25 residui C144, S2M11 raggiunge questo collegamento con un CDRH3 relativamente corto di 18 amminoacidi (definizione IMGT ( 48)). Di conseguenza, tutti e tre i leganti inibiscono SARS-CoV-2 interferendo con l’attaccamento dell’ACE2 a S attraverso la competizione diretta e il bloccaggio del trimero S nello stato chiuso. mAbs che riconoscono glicoproteine di superficie virali legandosi ad epitopi quaternari sono stati identificati contro il virus di Epstein-Bar ( 49 ), virus Dengue ( 50 – 53 ), virus Zika ( 54 ) virus Ebola ( 55 ), virus del Nilo occidentale ( 56 ) e HIV ( 57 ) e si è rivelato eccezionalmente potente o ampio. S2M11, insieme a Nb6 e C144, definisce quindi una classe distinta di potenti neutralizzatori di SARS-CoV-2 rispetto a mAb precedentemente isolati.
Abbiamo recentemente descritto che l’entità delle risposte degli Ab a SARS-CoV-2 S e nucleoproteine e titoli di Ab neutralizzanti è correlata ai punteggi clinici ( 28 ). Il SARS-CoV-2 RBD è l’obiettivo principale di potenti addominali S-specifici neutralizzanti nei sieri dei pazienti COVID-19 o nei campioni di plasma, concentrando così la maggior parte della pressione selettiva imposta dalla risposta immunitaria umorale su questo dominio ( 23 , 28). Dato che sono state trovate diverse varianti RBD tra gli isolati di SARS-CoV-2 circolanti, la combinazione di mAb specifici per RBD con diverse modalità di legame e distinti meccanismi di neutralizzazione potrebbe rivelarsi essenziale per un’applicazione clinica di successo. Una combinazione di S2M11 e S2E12 o cocktail di uno di questi mAb con S309 ha prodotto effetti additivi sulla potenza di neutralizzazione. Inoltre, i cocktail Ab comprendenti S309 e / o S2M11 hanno dimostrato una robusta attivazione di ADCC e ADCP, suggerendo che la combinazione di questi mAb utilizzando meccanismi di neutralizzazione distinti attiverebbe questi meccanismi protettivi in vivo. S2E12 e S2M11 (che ospitano un criceto Fc), singolarmente o formulati come cocktail, hanno conferito una protezione significativa utilizzando dosi di mAb che sono, a nostra conoscenza, le più basse segnalate per mAb umani testati su modelli di criceto. Di conseguenza,ci si aspetta che i cocktail mAb qui caratterizzati traggano vantaggio sia dalla neutralizzazione ultrapotente, sia da diversi meccanismi d’azione e dalle funzioni effettrici mediate da Fc per proteggere da un ampio spettro di isolati circolanti di SARS-CoV-2 e limitare l’emergere di mutanti di fuga dalla neutralizzazione. Proponiamo che le combinazioni di mAb che sfruttano più distinti meccanismi di azione con effetti additivi o sinergici potrebbero fornire ulteriori vantaggi per l’applicazione clinica.Proponiamo che le combinazioni di mAb che sfruttano più distinti meccanismi di azione con effetti additivi o sinergici potrebbero fornire ulteriori vantaggi per l’applicazione clinica.Proponiamo che le combinazioni di mAb che sfruttano più distinti meccanismi di azione con effetti additivi o sinergici potrebbero fornire ulteriori vantaggi per l’applicazione clinica.
Supplementary Materials
science.sciencemag.org/cgi/content/full/science.abe3354/DC1
Materials and Methods
Figs. S1 to S9
Tables S1 to S4
MDAR Reproducibility Checklist
This is an open-access article distributed under the terms of the Creative Commons Attribution license, which permits unrestricted use, distribution, and reproduction in any medium, provided the original work is properly cited.
References and Notes

