Dal COVID-19 al comune raffreddore: identificato un composto ampiamente efficace che blocca le infezioni
I ricercatori del Life Sciences Institute dell’UBC hanno identificato un composto che mostra una promessa iniziale nell’arrestare le infezioni da una serie di coronavirus, comprese tutte le varianti di SARS-CoV-2 e il comune raffreddore.
I risultati, pubblicati questa settimana su Molecular Biomedicine , rivelano un potenziale percorso verso trattamenti antivirali che potrebbero essere utilizzati contro molti agenti patogeni diversi.
“Oltre a COVID-19, ci sono molti diversi tipi di coronavirus che possono causare malattie gravi e talvolta fatali, e ancora di più è probabile che ne emergano in futuro”, afferma il dott. Yossef Av-Gay, professore di malattie infettive presso la facoltà di UBC di medicina e l’autore senior dello studio.
“Stiamo lavorando a trattamenti che possano essere ampiamente efficaci contro tutti i tipi di coronavirus in modo da poter rispondere non solo alle attuali sfide sanitarie, ma anche alle future minacce pandemiche. Identificare questo composto e il percorso attraverso il quale funziona per fermare i virus è un passo importante in quella direzione”.
Prendere di mira l’ospite, non il virus
I ricercatori attribuiscono l’ampia efficacia del composto al modo unico in cui funziona. Piuttosto che prendere di mira il virus stesso, il composto prende di mira un processo cellulare umano che i coronavirus utilizzano per replicarsi.
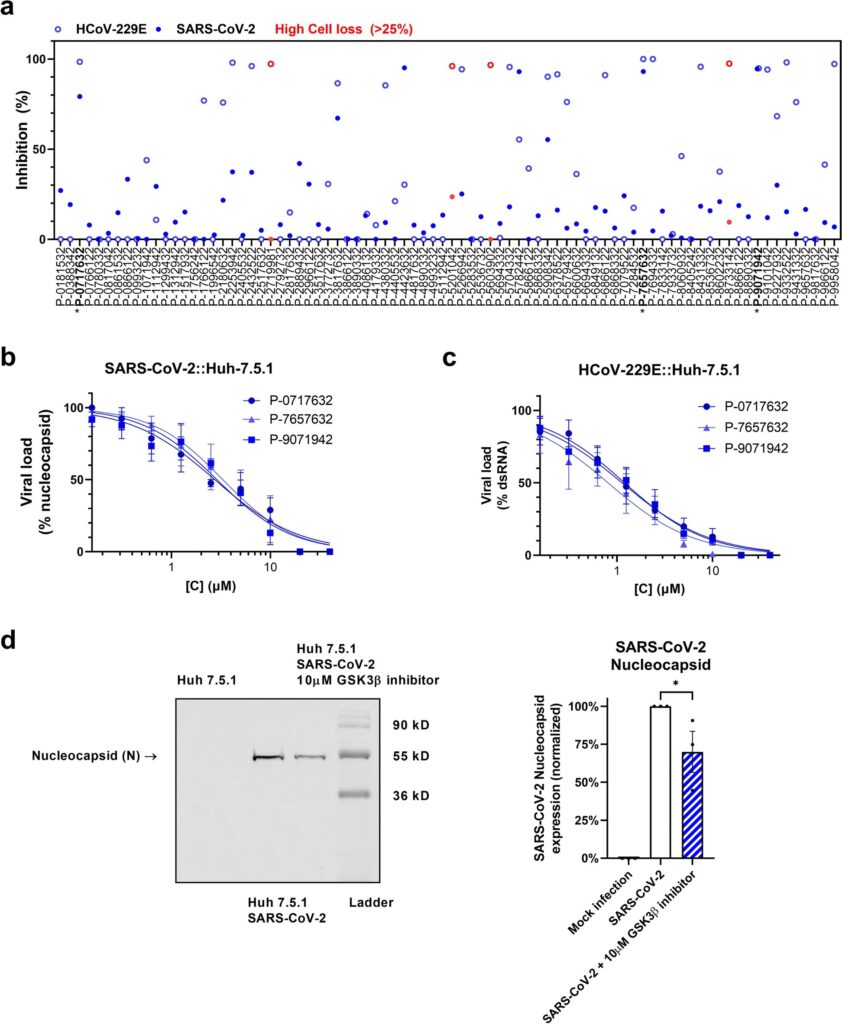
Poiché i virus non possono riprodursi da soli, si affidano ai percorsi di sintesi proteica nelle cellule ospiti per creare copie di se stessi. Nel caso dei coronavirus, usano un enzima umano chiamato GSK3 beta che esiste in tutte le cellule umane.
“Abbiamo scoperto che i coronavirus dirottano questo enzima umano e lo usano per modificare la proteina che racchiude il suo materiale genetico “, afferma il dott. Tirosh Shapira, borsista post-dottorato presso la facoltà di medicina dell’UBC e primo autore dello studio. “Questo composto blocca GSK3 beta, che a sua volta impedisce al virus di riprodursi e far maturare le sue proteine”.
Il composto fa parte di una più ampia famiglia di farmaci sperimentali noti come inibitori GSK3. Dalla fine degli anni ’90, scienziati di tutto il mondo accademico e industriale hanno studiato gli inibitori GSK3 per il loro potenziale come trattamenti per una serie di malattie, tra cui il diabete, l’Alzheimer e il cancro.
“Prendendo di mira questo percorso cellulare, piuttosto che il virus stesso, vediamo un’ampia attività contro più agenti patogeni. Stiamo anche agendo su un percorso che è finora immune ai cambiamenti tra varianti e diversi coronavirus”, afferma il dott. Shapira.
Trattamenti a prova di futuro
Per identificare il composto, il team di ricerca ha esaminato una libreria di quasi 100 noti inibitori GSK3, forniti attraverso una collaborazione tra UBC e Takeda Pharmaceutical Company in Giappone. I composti sono stati testati in modelli di cellule e tessuti infetti da SARS-CoV-2 e dal comune virus del raffreddore.
Il test ha prodotto più inibitori GSK3 che hanno mostrato un alto livello di efficacia contro i coronavirus e una bassa tossicità per le cellule umane. Il composto principale, identificato come T-1686568, ha inibito sia SARS-CoV-2 che il comune virus del raffreddore , i criteri principali utilizzati dagli autori nella ricerca di una protezione ad ampio spettro.
“Anche se questi sono i primi giorni, è incoraggiante vedere ampi livelli di efficacia nei modelli di tessuto”, afferma il dott. Shapira. “Poiché questi composti richiedono molti anni di test e approvazione normativa prima che possano potenzialmente raggiungere i pazienti, dobbiamo pensare ad applicazioni a lungo termine e a come ciò potrebbe applicarsi ampiamente a futuri virus e varianti”.
La ricerca è stata condotta presso UBC FINDER, una struttura di biocontenimento di livello 3 presso UBC, dove i ricercatori stanno lavorando con agenti patogeni altamente infettivi con l’obiettivo di sviluppare trattamenti futuri.
“Non stiamo solo combattendo SARS-CoV-2, stiamo guardando avanti a ciò che verrà dopo”, afferma il dott. Shapira. “Siamo concentrati sull’identificazione di trattamenti a prova di futuro per varianti e virus che emergono lungo la strada e si basano sugli stessi meccanismi cellulari per crescere e infettare”.

